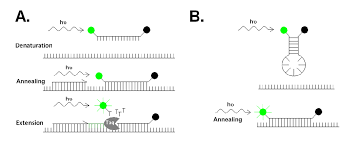
Real-time PCR نوعی از آزمایش PCR استاندارد است که طی آن، میزان محصولات تولیدشده همزمان با انجام فرایند بررسی میشود.دستگاه ریل تایم پی سی آر (Realtime PCR) با هدف تعیین میزان بیان ژن های موجودات زنده راه اندازی و ارتقا داده شده است.آشنایی با این روش و یادگیری مهارت های لازم یکی از تکنیک های بسیار پر کاربردی است که باعث می شود شما به سرعت وارد بازار کاری در رشته های علوم زیستی و آزمایشگاهی شوید.
ریل تایم پی سی آر (Realtime PCR)
دستگاه ریل تایم پی سی آر (Realtime PCR) با هدف تعیین میزان بیان ژن های موجودات زنده راه اندازی و ارتقا داده شده است. روش پی سی آر (PCR) ساده دارای نقص و محدودیت هایی از قبیل عدم توانایی مطالعه ی کمی میزان بیان یک ژن میباشد.
بنابراین سیستمی توسعه یافته با عنوان ریل تایم پی سی آر (Realtime PCR) ارائه شده است که هدف اصلی از این سیستم نشان گذاری ژن های موجودات زنده می باشد که مسیر کل مجموعه ژنومی منتهی به ساخت محصولاتی از جنس RNA و پروتئین میشود. در واقع RNA یا ریبونوکلویک اسید های یک سلول می تواند قادر به تعیین خصوصیت و وجه تمایز یک سلول در سطح فنوتیپی دارد و نیز نشان میدهد در چه سطحی آن خصوصیت را بروز می دهد .
از طرفی بجهت اینکه DNA همیشه ثابت بوده و یک ژن مشخص در بین موجودات یک گونه شباهت بسیاری به هم دارد و هیچ نشانه ای از میزان بیان آن ژن ارائه نمی دهد عمل مقایسه دو سلول از نظر خصوصیات و فنوتیپی را نمی توان در سطح DNA بررسی نمود،.بنابراین مطالعه ی بیان یک ژن می تواند در سطح RNA و یا پروتئین انجام پذیرد. همچنین بسیاری از ژن ها در نهایت به RNA تبدیل شده و نقششان را باعنوان RNA به انجام می رسانند. در نتیجه در برخی موارد محصول ژنی به شکل RNA عرضه شده و هیچ گاه قرار نیست به پروتئین ترجمه شود. در اینصورت برای بیان آن ژن مشخص که فقط رونویسی می شود ولی ترجمه نمی شود، فقط RNA مورد بررسی می باشد.
مولکول RNA یا ریبونوکلویک اسید به دلیل داشتن اکسیژن در کربن شماره 2 و همچنین تک رشته و کوتاه بودن آن، نسبت به مولکول های DNA واکنش پذیر تر بوده و تخریب پذیری بالایی دارد.
در ریل تایم پی سی آر (Realtime PCR) جهت بررسی بیان یک ژن، ابتدا با کمک آنزیم ترانس کریپتاز معکوس (آنزیم MMLV) مولکول های RNA را سنتز و به DNA های مکمل که غالبا cDNA نامیده می شوند، تبدیل میکنند. پس از پایداری مولکول مورد سنجش، وارد سیکل های ریل تایم پی سی آر (Realtime PCR) شده و بیان مورد مطالعه قرار می گیرد. دستگاه ریل تایم پی سی آر (Realtime PCR) از یک سیستم اپتیکی بهره برده که توسط آن میزان DNA هر نمونه محاسبه می گردد. زمانی که cDNA سنتز شد، پس از آماده سازی نمونه جهت ورود به سیکل های پلیمریزه شدن، به نمونه DNA رنگی تحت عنوان سایبرگرین (SYBR Green) اضافه می گردد. این محصول موجب رنگ شدن DNA شده و همچنین خود دارای ویژگی فلورسانتی می باشد. زمانی که ریل تایم پی سی آر (Realtime PCR) سیکل های پلیمریزاسیون خود را شروع کرد، به ازای ساخته شدن رشته های DNA جدید، رنگ سایبر گرین به رشته متصل می شود و دستگاه ریل تایم پی سی آر (Realtime PCR) توسط فناوری منحصربفرد خود خوانش فلورسانتی را شروع می کند. لذا روش ریل تایم پی سی آر (Realtime PCR) با ویژگی فلورسنتی سایبر گرین (SYBR Green) برای هر نمونه یک گراف تکثیر ترسیم می کند. هر نقطه از گراف تکثیر نشان دهنده میزان اتصال SYBR Green به رشته DNA می باشد و در نهایت گراف که در نمایشگر متصل شده به ریل تایم پی سی آر (Realtime PCR) به نمایش در آمده، به آستانه خود می رسد. این میزان نشان دهنده میزان DNA بیان شده در هر نمونه می باشد.
تمامی اصول و واکنشگرهایی که برای یک PCR معمولی نیاز است در تکنیک Real time PCR ( ریل تایم پی سی آر ) هم بکار میرود، اما یک گزارشگر فلورسنت نیز در واکنش حضور دارد. این گزارشگرها به گونهای طراحی میشوند که در صورت تکثیر DNA نور تولید کنند. لذا افزایش شدت نور ثبت شده در دستگاه با میزان محصول بدست آمده نسبت مستقیم دارد. با ادامه یافتن PCR شدت فلورسنت رو به افزایش میگذارد.
به اولین چرخهای که شدت فلورسنت بیشتر از خط پایه باشد چرخۀ آستانه یا CT گویند. عدد CT با مقدار الگوی اولیه رابطۀ معنیدار دارد و از روی آن میتوان مقدار mRNA اولیه را مورد تخمین قرار داد.
به عبارت دیگر در فاز اولیه مرحله تصاعدی مقدار فلورسنت افزایش می یابد تا به آستانهای می رسد، که به مقدارمشخصی از سطح background بالاتر است، این چرخه ( سیکلی ازPCR که قطعه تکثیر از حد آستانه عبور می کند) به عنوان CT شناخته می شود.
آشنایی با مفهوم دلتا Ct (∆Ct) در آنالیز و تفسیر نتایج تکنیک ریل تایم (real Time PCR)
با توجه به اینکه نتایج ریل تایم ارائه شده در شکل 6 دارای همه شرایط فوق می باشد به آنالیز داده های شکل فوق بپردازیم. در این شکل همان گونه که مشاهده می شود بین Ct های ژن کنترل داخلی RPLP0 در بین نمونه های سرطانی و نرمال یک Ct تفاوت وجود دارد. بنابراین باید این تفاوت را در مقایسه Ct ژن YHZ2 بین نمونه سرطانی و کنترل اعمال نماییم و به اصطلاح داده ها (که همان Ctها هستند) را نرمال نماییم ( این نرمال نمودن به معنی نرمال یا سرطانی بودن نیست). برای نرمال کردن داده ها، Ct ژن کنترل داخلی را همه نمونه ها از Ct ژن مورد بررسی کم می کنیم. از آنجایی که داده های نرمال برای هر نمونه از کم نمودن Ct ژن کنترل داخلی از Ct ژن مورد نظر از به دست آمده است داده نرمال شده را دلتا Ct (∆Ct)می نامیم (شکل 7).
مزایای روش ریل تایم (Real time PCR)
☑️ امکان رویت لحظه به لحظه یک واکنش مهیا است و در هر دوره ( سیکل) ، کاربر میتواند پروسه تکثیر را بررسی کند. به عبارت دیگر و بطور خلاصه تکثیر در هر زمان قابل رویت و بررسی است.
☑️ امکان شرایط ارتقا واکنش با مناسبترین غلظت DNA ، پرایمر و تعداد سیکل ها (دوره ها) برای تکثیر امکان پذیر است.
☑️ در هر زمان که کاربر تشخیص دهد میتواند برای جلوگیری از اتلاف وقت و انرژی ، واکنش را پایان دهد چراکه روند PCR قابل رویت و بررسی است و عدم تکثیر و رفتن به حالت سکون کاملا مشخص است.
☑️ میزان دقیق و نسبی الگوی اولیه قابل شناسایی است.
☑️ واکنش ها سریع تر و در زمان کوتاه تری انجام میشود.
☑️ محدوده تشخیص آن بیشتر از PCR معمولی است بطوریکه اختلاف کمتر از 2 برابر هم نشان میدهد.
مراحل کار Real time PCR ( ریل تایم پی سی آر )
☑️ فاز اول The baseline region ; با وجود اینکه محصول دو رشته ای وجود دارد ولی نور آن قابل ردیابی نیست.
☑️ فاز دوم The exponential phase ; محصول دو رشته ای در هر چرخه دو برابر می شود و رشد نمایی مربوط
به واکنش شروع می شود.
☑️ فاز سوم The liner phase; ترکیبات واکنش و کارایی آنها روبه اتمام است.
☑️ فاز چهارم The plateau phase; ترکیبات واکنش از بین می روند و افزایش در میزان فلورسنت مشاهده نمی شود.در پروتکل انجام Real time PCR ( ریل تایم پی سی آر )واکنش بصورت تک مرحله ای ویا ۲ مرحله ای انجام میشود.
در واکنش یک مرحله ای سنتز cDNA و تکثیر آن در یک تیوب انجام می شود. در واکنش ۲مرحله ای نسخه برداری معکوس و تکثیر در تیوب های جداگانه صورت می گیرد. در روش تک مرحله ای تغییرات آزمایشگاهی به حداقل می رسد ولی حساسیت کمتری دارد. در روش ۲مرحله ای هم امکان سنجش قسمتهای مختلف یک DNA هدف وجود دارد و حذف پرایمر دایمر با دستکاری دمای Annealing آسانتر است گر چه ممکن است شانس آلودگی را بیشتر کند ولی دارای مزیت های بیشتری نسبت به واکنش تک مرحله ای است.
روشهای شناسایی PCR Real time
در سالیان اخیر پیشرفتهای تکنیکی زیادی اتفاق افتاده و ابزار زیادی در این روش بکار رفته که مهمترین آن شاید اختراع دستگاههای چند کاناله باشد. این دستگاهها قادرند به صورت همزمان چندین طول موج نوری متفاوت را تابانیده و بازتابش آن را ثبت نمایند. دراین تکنیک ، برای تعیین غلظت DNA از رنگهای فلورسانس و یا شاخص های الیگونوکلئوتیدی فلورسانس استفاده میشودکه به برخی از مهمترین آنها اشاره میشود:
رنگهای فلورسنت متصل شونده بهDNA
شاید متداول ترین رنگ مورد استفاده در این تکنیک سایبرگرین I باشد. این رنگ اینترکاله وفلورسنت، به شیارهای کوچک DNA دورشتهای متصل میشود و باجذب طول موج ۴۹۸ نانومتر، نور ۵۲۲ نانومتری را ساطع میکند که توسط دستگاه ثبت میشود.
سایبرگرین I به الگوهای تک رشتهای متصل نمیشود لذا در این موارد بازتاب ضعیفی دارد. در طی چرخه های PCR که محصول دو رشته ای تولید میشود، رنگ به آنها متصل می شود و بنابراین افزایش شدت نور فلورسنت با غلظت dsDNA متناسب است.
سایبرگرین I یک رنگ غیر اختصاصی است لذا برای تمامی آزمایشات قابل استفاده است و این مسئله یک مزیت به شمار میآید.
از دیگر مزایای این رنگ میتوان به ارزانی، عدم تداخل با پلیمرازها، ساده و غیر سمی بودن آن اشاره کرد. همانگونه که گفتیم این رنگ غیر اختصاصی است و این مزیت، مهمترین عیب آن را نیز شامل میشود چرا که نمیتوان وجود محصولات غیر اختصاصی یا پرایمر دایمر را به کمک این رنگ شناسایی کرد. البته استفاده از نمودار منحنی ذوب در دستگاههای امروزی این مشکل را تا حدودی مرتفع کرده است.
پروبهای فلورسنت
پروبها بر خلاف سایبرگرین I بر اساس تشخیص اختصاصی توالی محصول کار میکنند. پروبها معمولاًیک رنگ فلورسنسزا (Reporter) و یک رنگ خاموش کننده (Quencher) دارند. معمولاً دستگاهها بازتابش رنگ فلورنسزا را بررسی مینمایند حضور خاموش کننده در نزدیکی موقعیت فلورسنسزا موجب جذب نور آن و خاموش شدن آن میشود لذا در این حالت بازتابشی در دستگاه ثبت نمیشود.از این پروب ها در روشهای زیر استفاده می شود.
Hydrolysis probe
نمونه بارز این روش پروبهای Taqman هستند همچنین این روش به عنوان سنجش’۵ نوکلئازی نیزشناخته می شود. پروب فلورسنت هیدرولیز شده ودر نتیجه در محصول PCR تشخیص داده می شود. پروب با ترادف اختصاصی با رنگ گزارش دهنده (reporter) در انتهای’۵ و خاموش کننده (quencher) در انتهای ‘۳ قرار داده می شود.
روش استفاده از پروبهای TaqMan بر دو حقیقت استوار است اول آن که از یک آنزیم DNA پلیمراز با ویژگی اگزونوکلئازی ‘۵ به ‘ ۳ استفاده شود و دیگر این که پروب اولیگونوکلئوتیدی حاوی دو نشان رنگی(یکی فلورسنسزا و دیگری خاموش کننده) است و تنها زمانی سیگنال میدهد که بافعالیت اگزونوکلئازی شکسته شود. پروب مورد نظر از طریق توالی اختصاصی به محصولات
متصل میشود.
Hybridization probe
در اینجا دو پروب وجود دارد یک پروب با یک فلورفور دهنده و پروب دیگر با یک رنگ گیرنده در اتصال است. در حین واکنش PCR پروب ها طوری به الگو هیبرید می شوند که از سر دم در اتصال قرار می گیرند. در این جهت انرژی پروب دهنده رنگ پروب گیرنده را تحریک می کند که منجر به ساطع شدن نور با طول موج بالاتری می شود. نسبت بین فلورسنس دهنده به فلورسنس گیرنده در طول واکنش افزایش می یابد که متناسب با DNA تکثیری است، اگر DNA تکثیر نشود هیچنوری قابل ساطع نیست زیرا پروب ها به DNA متصل نشده اند.
Hairpin probe
بیکونهای مولکولی (Molecular beacons)سادهترین پروبهای سنجاق سری هستند که شامل یک ناحیه با ترادف اختصاصی (ناحیه لوپ) که دو طرف آن توالیهای معکوس تکراری وجود دارد. رنگهای Reporter و quencher به دو انتهای مولکول متصل میشوند. این دو وقتی با هم در تماس هستند، انرژی انتقالی رزنانسی آنها کاهش مییابد.
پروبها با ساختار سنجاق سری منجر به اختصاصیت بیشتر نسبت به پروبهای خطی میشوند. کمپلکس Probe-Target باید از لحاظ ترمودینامیکی پایداری بیشتری نسبت به ساختار سنجاق سری پروب به تنهایی داشته باشند. زیرا در کمپلکس Probe-Target ترکیب پایدارتری با استفاده از همولوژی بازها تشکیل میشود.
این ویژگی به خصوص در تشخیص آللها بکار میرود. مهمترین مزیت بیکنهای مولکولی این است که در جریان واکنش دست نخورده باقی میمانند و در چرخههای بعدی مجدداً هیبرید میشوند. میزان تشعشع فلورسنس درمرحلۀ هیبریداسیون، با غلظت ژن هدف متناسب است .
منحنی ذوب
برای اینکه بفهمیم PCR درست کار کرده است باید از منحنی ذوب استفاده کنیم. به این ترتیب که از دمای پایین تر از دمای اتصال تا دمای بالاتراز ۹۵ درجه سانتی گراد را در نظر گرفته وبرنامه مربوط به آن را به دستگاه می دهیم، تا به ازای هر نیم درجه سانتی گراد لامپ روشن شود و شدت جذب فلورسنت را قرائت کند این باعث می شود که برای تمام قطعات تکثیر شده پیک جذب رسم شود. به این ترتیب در صورت وجود آلودگی از طریق دیدن پیک جذب آن را شناسایی می کنیم. در دمایی پایین پیک های که رسم می شود مربوط به دایمر آغازگر است و آخرین پیک مربوط به قطعه موردنظر ماست.
آنالیزهای کمی درReal time PCR
دو روش عمده برای بررسی کمی در time PCR Real وجود دارد:
☑️ روش منحنی استاندارد (مقایسۀ مطلق)
در این روش از نمونۀ RNA یا DNA با غلظت مشخص برای رسم منحنی استاندارد استفاده میشود. غلظت RNA یا DNA استاندارد با اسپکتروفوتومتر (nm260) تعیین میشود و سپس از روی وزن مولکولی نمونه به تعداد نسخههای آن تبدیل میشود.
استانداردهای غلظتی ژنهای معروف به صورت تجاری قابل خریداری است هرچند که بسیار گران قیمتند. از نمونه های استاندارد سری رقعت تعیین کرده و همراه با نمونه هدف در دستگاه Real-time PCR قرار می دهیم با استفاده از Ct که دستگاه برای هر رقت به ما می دهد یک منحنی رسم کرده که X آن رقت یا تعداد کپی از ژن و Y آن CT باشد، نمودار به دست امده یک نمودار خطی است که با قرار دادن عدد نمونه هدف در نمودار غلظت یا تعداد کپی آن نیز بدست می آید. ترجیحا طول قطعه استاندارد مساوی با طول قطعه هدف باشد.
☑️ روش آستانه نسبی (مقایسۀ نسبی)
برای حذف نوسانات مقادیر RNA وارد شده در واکنش و خطاهای عملکرد دستگاهها و فرد از استانداردهای داخلی استفاده میشود. این استانداردها باید در همۀ بافتها بیان ثابت داشته باشند و آزمایش ما نباید بیان آن را نسبت به نمونۀ کنترل تغییر دهد. بدین منظور از ژن بتا اکتین و GAPDH استفاده میشود. به این ژنها Housekeeping گویند. بیان این ژن ها ثابت است که با مقایسه نمونه تیمار شده با شاهد و کنترل داخلی می توان کاهش یا افزایش بیان را مشاهده کرد.
تشخیص پلی مرفیسم توسط Real time PCR
استفاده از این تکنیک اجازه تعیین کمیت نواحی چند شکلی DNA و یا تعیین ژنوتیپ پلیمرفیسم یک نقطهای SNPS) Single nucleotide polymorphism,) جدید را به ما می دهد. مانیتور کردن همزمان پروسه تکثیر و تعیین ژنوتیپ با استفاده از هیبریداسیون پروبها و با آنالیز منحنی ذوب امکان پذیر می شود.
موتاسیون نقطه ای بوسیله آنالیز منحنی ذوب تشخیص داده میشود. دمای بحرانی Tm که در آن DNAتک رشته ای میشود یک شناساگر برای وجود یا عدم وجود موتاسیون میباشد.
پروب اختصاصی برای ناحیه موتانت تهیه می شود در دمای پایین پروب به ناحیه های غیر اختصاصی متصل می گردد و با افزایش دما پروب اختصاصی تر عمل می کند، منحنی ذوب حاصل از این واکنش را آنالیز می کنیم تا متوجه وجود یا عدم وجود موتاسیون شویم.
آشنایی با مفهوم Fold change و نحوه محاسبه دلتا دلتا Ct (∆∆Ct) در ریل تایم
در واکنش ریل تایم هنگامی که میزان بیان یک ژن را در چند گروه مقایسه می نماییم بهترین راه بیان میزان تغییرات آن ژن در یک نمونه نسبت به نمونه دیگر fold change می باشد. مفهوم fold change همان “چند برابر” می باشد، به عبارت بهتر بیان ژن مورد نظر در نمونه های گروه اول (به عنوان مثال نمونه های سرطانی) نسبت به نمونه های گروه دوم (به عنوان مثال نمونه های نرمال) چند برابر افزایش و یا کاهش یافته است؟ این جمله به این مفهوم است که اگر بیان ژن مورد بررسی را در نمونه نرمال برابر 1 در نظر بگیریم، بیان آن در نمونه سرطانی برابر چه میزانی خواهد بود؟
قبل از توضیح محاسبه fold change در real time به این مثال توجه فرمایید وزن فرد A برابر 60 کیلوگرم و وزن فرد B برابر120 گیلوگرم است، پس Fold change وزن فرد B نسبت فرد A برابر است با 2 برابر (120 تقسیم بر 60). توجه داشته باشید که در اعلام نتایج باید Fold change فرد A نسبت خودش هم ذکر شود زیرا برای بررسی های آماری مورد نیاز می باشد
آموزش نحوه بررسی کیفیت نتایج ریل تایم
قبل از آنالیز داده ها در ابتدا باید کیفیت نمونه ها در چند مرحله چک شوند.
مرحله اول: در بیشتر نمونه ها Ct ژن bcl1 از 38 کمتر می باشد. توجه کنید که در بیشتر نمونه های هر دو گروه نرمال وسرطانی نباید Ct ژن bcl1 از 38 بیشتر باشد. چون Ct بالای 38 درReal time قابل قبول نمی باشد. اما اگر در گروه نرمال همه Ctها بالای 38 بود اما در گروه سرطانی اکثرا زیر 38 باشد این Ct ها قابل قبول هستند زیرا می توان گفت این ژن در گروه نرمال بیان ندارد اما در گروه تومری بیان دارد ( یا برعکس).
مرحله دوم: برای هر دو ژن bcl1 و gapdh در هر دو تکرار برای یک نمونه، نباید اختلاف Ct بیش از 1 باشد. در جدول 1 اختلاف Ct ژن gapdh در تکرارهای نمونه شماره 19 برابر 1.74 می باشد بنابراین این نمونه باید از آنالیز ها بیرون گذاشته شود (به شکل زیر توجه کنید).
شکل فوق: در این شکل 10 نمونه بررسی شده اند که با رنگ های مختلف نشان داده شده اند. هر نمونه 5 بار تکرار شده است و مشاهده می شود که منحنی همه تکرارها برای هر نمونه برروی هم منطبق می باشند. اما در صورتی که یک تکرار منحنی آن با تکرار های دیگر منطبق نباشد باید آن تکرار از بررسی ها حذف شود.
آموزش تفسیر منحنی ذوب (melt curve) در ریل تایم پی سی ار
مرحله سوم: بررسی منحنی ذوب (melt curve) نمونه ها. در صورتی که از رنگ های فلورسنت مانند سایبرگرین در ریل تایم استفاده نموده اید می توانید منحنی ذوب را برای هر نمونه مورد بررسی قرار دهید. از آن جاییکه هر ژن دارای منحنی ذوب خاص خود می باشد بنابراین منحنی های یک ژن در تمام نمونه ها باید با هم منطبق باشند و همچنین باید تمام منحنی ها تک قله باشند. به شکل های زیر توجه کنید.
در آنالیز داده های ریل تایم نمونه هایی که منحنی ذوب آن ها برای هر دو ژن با سایر نمونه ها منطبق نیست باید از بررسی حذف شوند. توجه کنید که منحنی ذوب ژن bcl1 و ژن کنترل داخلی باید جدا گانه مورد بررسی قرار گیرند و نیازی نیست منحنی های این دو ژن با هم منطبق باشد.
بعد از حذف نمونه های غیرقابل قبول نوبت به آنالیز داده ها می رسد در این مرحله چون تمام تکرارها در یک واکنش Real time به صورت هم زمان بررسی شده اند باید از Ct های حاصله برای هر نمونه میانگین گرفت و با ct میانگین کار کرد.
نکته: در مرحله میانگین گرفتن توجه شود که، در این مثال در یک واکنش ریل تایم، هر نمونه 2 بار همزمان تکرار شده است بنابراین باید برای هر نمونه میانگین گرفت. اما اگر نمونه ها چند نوع باشند به عنوان مثال یک تیمار که دو بارتکرارشده است، دیگر نباید بین تکرار تیمارها میانگین گرفت.
نکته: در صورتی که یک ریل تایمی که قبلا اجرا شده دو باره تکرار شود. نباید بین Ct نتایج قدیم و جدید میانگین گرفته شود. نحوه آنالیز این داده ها در ادامه توضیح داده خواهد شد.
سرفصل های آموزشی در دوره Real-Time PCR
☑️ استخراج RNA و سنتز cDNA به صورت عملی
☑️ اصول real-time PCR
☑️ روشهای انجام real-time PCR
☑️ انواع پروبها و کاربردهای آتها
☑️ اصول طراحی پرایمر و پروب
☑️ مقایسه روشهای نسبی (relative) و مطلق (Absolute) و کاربردهای آنه
☑️ انواع ژنهای رفرانس درروش نسبی
☑️ آماده سازی مواد و master mix
☑️ کار عملی با دستگاه Real-time PCR
☑️ کار با نرم افزار و آنالیز داده ها
☑️ Troubleshooting
شما می توانید مطالب زیر را مطالعه کنید:






















.jpg)

.jpg)
.jpg)


.jpg)
.jpg)